서 론
Small heterodimer partner(SHP)-interacting leucine zipper protein(SMILE)은 처음에 Zhangfei라고 불리는 basic leucin zipper protein(bZIP) 전사조절인자로, 바이러스 단백질 VP16을 통해 단순포진바이러스(Herpes simplex virus, HSV)의 극초기 유전자(immediate early genes) 전이 활성화를 위해 필요한 수용체 세포 인자(host cell factor, HCF)에 결합하는 보조조절인자(coregulator) 중 하나로 알려졌다[1,2]. bZIP 단백질은 이합체화(dimerization), DNA 결합, 전사조절을 담당하는 세 개의 기능 부위가 있는 반면에, SMILE은 bZIP 단백질과 달리 bZIP 결합 부위나 프로모터 결합 부위에 결합하지 못한다[1,3]. bZIP 단백질은 류신과 아르기닌이 많은 구조로 이루어져 있으며, 이러한 구조는 DNA 넓은 홈(major groove)에 결합하여 전사활동을 촉진한다[4-6]. 또한, bZIP 아미노산 잔기 중 아스파라진, 아르기닌, 류신 및 이소류신 잔기가 DNA 결합에 중요하게 작용한다고 보고된 바가 있다[7]. 실제로, bZIP 단백질 중 하나인 C/EBP-homologous protein-10은 아스파라진 잔기가 없어, DNA에 결합하지 않았다[8]. SMILE 또한 마찬가지로 아스파라진 잔기의 결핍에 의한 구조적 변화로 DNA에 결합할 수 없다는 것을 보여주었다[9]. 이러한 결과는 SMILE이 bZIP 단백질과 달리 DNA에 결합하는 전사조절인자가 아닌 보조조절인자로 작용한다는 것을 시사한다.
추가 연구를 통해 SMILE은 다양한 핵수용체 및 전사조절인자의 보조조절인자로 밝혀졌다[10]. 핵수용체는 리간드 비의존적인 전사 활성 도메인(Activation Function 1, AF-1), DNA 결합 도메인(DNA binding domain), hinge 도메인, 리간드 결합 도메인(ligand binding domain, LBD), 리간드 의존적인 전사 활성 도메인(AF-2) 5개의 도메인으로 이루어져 있다. 핵수용체는 표적유전자의 프로모터 전사조절부위에 직접 결합하여, 보조활성인자(coactivator) 혹은 보조억제인자(corepressor)와의 상호작용을 통해 유전자 발현을 조절하는 리간드 의존성 전사조절인자이다[11]. 리간드가 없을 때 SMILE은 핵수용체 전사활성을 저해하는 보조억제인자로 작용하며, 실제로 constitutive androstane receptors(CAR), retinoid X receptors(RXR), and estrogen-related receptor gamma(ERRγ) 등 여러 핵수용체의 전사활성을 저해한다[10,12]. 따라서, 다양한 대사과정에 관여하는 bZIP 단백질 또는 핵수용체와 SMILE의 상호작용을 이해하는 것은 매우 중요하다.
흥미롭게도, SMILE은 세포 외부 자극에 따라 유전자 발현이 조절되는 보조억제인자며, 간에서 인슐린(insulin), 메트포민(metformin), 담즙과 같은 내분비 대사신호의 mediator로 작용한다고 알려져 있다[13-15]. 그렇기 때문에, SMILE은 당뇨, 비알콜성지방간(non-alcoholic fatty liver disease, NAFLD), 고혈당, 낭포성 섬유종 같은 질병을 억제하는데 중요하다[14,16]. 최근 연구들을 통해 SMILE은 포도당 대사, 지질 대사, 담즙 대사와 관련된 liver X receptor(LXR), pregnane X receptors, farnesoid X receptor, CAR, hepatocyte nuclear factor 4(HNF4), and ERRs과 같은 핵수용체와, cAMP response element-binding protein(CREB), CREBH, steroid regulatory element binding protein-1c(SREBP-1c), forkhead box protein O1(FoxO1), and signal transducer and activator of transcription 3 같은 전사조절인자의 전사활성을 억제하는 것으로 알려졌다[10,17-19]. 이러한 이유들을 통해, SMILE이 대사관련 질병의 중요한 표적유전자가 될 수 있다는 것을 시사한다.
본 론
SMILE은 bZIP 단백질의 CREB/activator transcription factor(ATF) 계열의 단백질이다[20]. SMILE은 estrogen receptor(ER)에 의한 전사조절을 저해하는 SHP에 결합하여 SHP의 억제작용을 증가시키는 것이 yeast hybrid 시스템을 이용하여 밝혀졌다[21]. 이러한 결과를 통해 SMILE이 핵수용체의 전사조절을 억제하는 보조억제인자로 작용한다는 것을 보여주었다. 구조적으로, SMILE은 354개의 아미노산으로 이루어져 있으며, 이 중 113부터 202번째 아미노산은 핵수용체 LBD와 결합하기 때문에 그것의 보조억제인자 활성에 중요하다. SMILE은 선택적 스플라이싱(alternative splicing)을 통해 두 개의 동형체(isoform)를 가지고 있으며, 긴 동형체(SMILE-L)는 짧은 동형체(SMILE-S)와 비교하여 83개의 아미노산을 추가로 가지고 있다(Fig. 1A). SMILE은 특정 전사조절인자와 상호작용에 의해 보조활성인자 혹은 보조억제인자로 작용한다. 예를 들면, ATF4와 heterodimer 복합체를 이루어 CRE reporter의 활성을 증가시키는 반면에, CREB3와 결합하여 HCF 활성을 억제하는 보조억제인자로 작용한다[22]. 흥미롭게도, 간에서 SMILE은 보조억제인자로 존재하며, curcumin, ursodeoxycholic acid(UDCA), epigallocatechin gallate-1, 메트포민, 인슐린과 같은 인자들에 의한 신호전달경로에 의해 유전자 발현이 조절된다[18,23,24]. 이러한 인자들에 의해 매개된 SMILE은 다른 전사조절인자 혹은 핵수용체의 상호작용을 통해 활성을 저해하여 대사, 발달, 항상성 등을 조절한다.
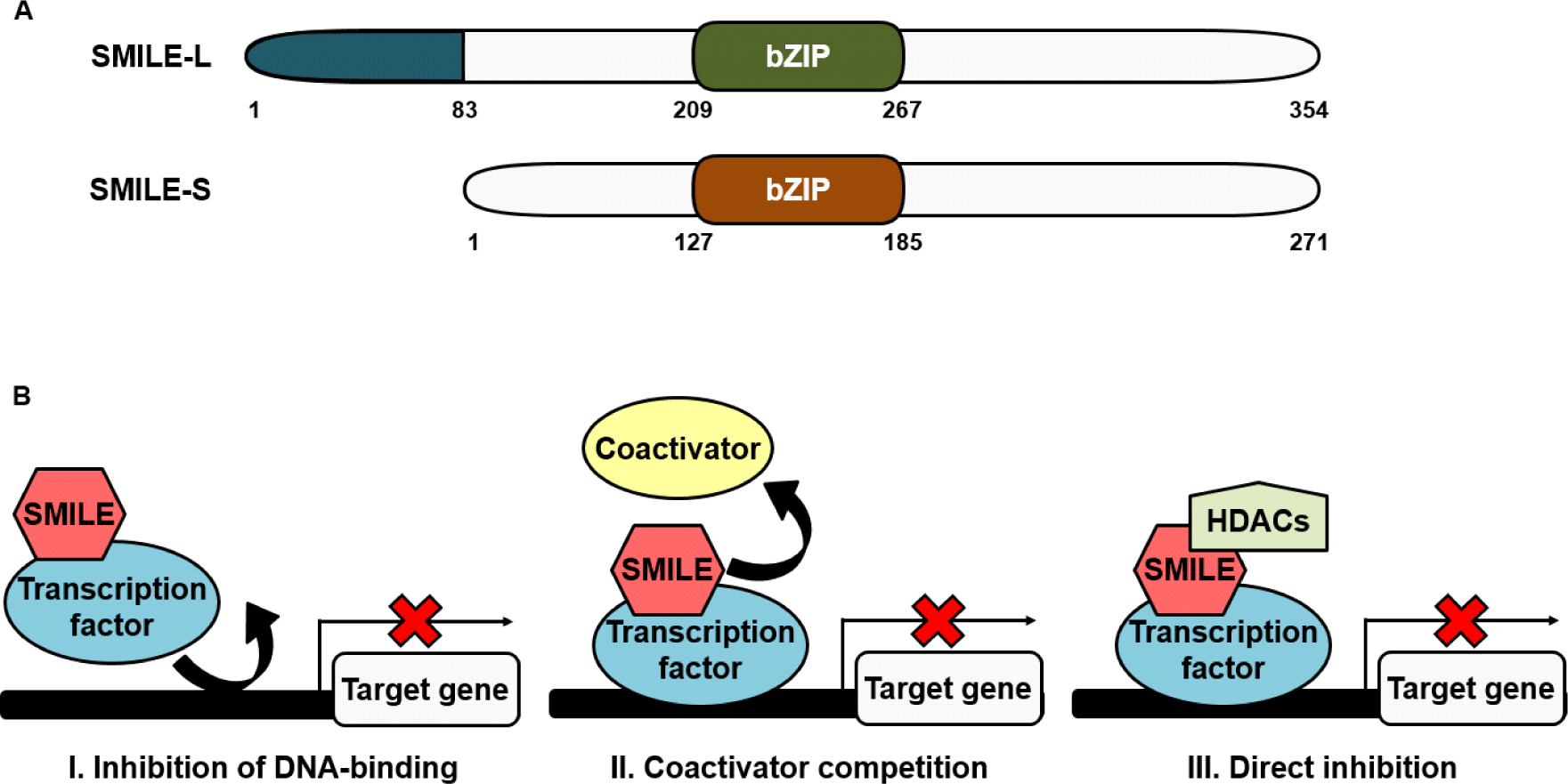
SMILE은 다양한 방법으로 핵수용체와 bZIP 전사조절인자의 활성을 조절한다(Fig. 1B). 첫째로, peroxisome proliferatffor-activated receptors(PPARs), HNF4, LXRs, CAR, ERRγ, runt-related transcription factor 2와 같은 핵수용체와 전사조절인자의 프로모터 결합을 억제하여 전사활성을 저해한다. PPARs은 thyroid hormone receptor과 유사한 핵수용체이며, RXR과 heterodimer를 구성한다. PPARs은 리간드가 존재할 때 보조활성인자와 복합체를 이루어 유전자 발현을 조절하지만, 리간드가 없을 때 보조억제인자가 PPAR과 RXR heterodimer에 결합해 complex의 DNA 결합을 억제한다. PPARγ는 adiponectin(Adipoq) 프로모터에 직접 결합하고, 지방세포 분화를 촉진시켜 지방축적을 유도한다[25]. 실제로, ER-stress에 의해 유전자 발현이 유도된 SMILE은 PPARγ에 직접 결합하고, PPARγ의 Adipoq 프로모터 결합을 억제하여 유전자 발현을 감소시켜 지방 축적이 줄이는 것으로 보고되었다[26]. 둘째로, SMILE은 핵수용체의 LBD 혹은 AF-2 도메인과 결합하여 전사활성을 조절하며, proliferator-activated receptor γ coactivator-1α(PGC-1α), glucocorticoid receptor interacting protein 1과 같은 보조활성인자와 경쟁을 통해 전사활성을 억제한다[10]. PGC-1α는 HNF4α, FoxO1, glucocorticoid receptor와 같은 전사조절인자에 결합하여 glucose-6-phosphatase(G6Pase) 및 phosphoenolpyruvate carboxykinase(PEPCK)와 같은 당신생합성(gluconeogenesis) 유전자 발현을 조절한다고 알려져 있다[27]. 실제로, SMILE은 HNF4α과 PGC-1α의 결합을 방해하여 당신생합성 유전자의 발현을 억제한다[10,28]. 마지막으로, SMILE은 histone deacetylases(HDACs)과 상호작용하여 전사조절인자에 의한 표적유전자의 발현을 직접 억제한다. HDAC은 히스톤의 아세틸그룹을 제거하여 히스톤 꼬리와 DNA의 상호작용을 강화하고 DNA에 전사 유도 단백질 복합체의 접근을 차단시켜 전사과정을 억제한다[29]. 실제로, GST-pull down assay를 통해 SMILE은 HDAC(HDAC1, HDAC2, HDAC3, HDAC4, HDAC5, and HDAC6)중 HDAC1, 3, 4와 결합하는 것으로 밝혀졌다[10]. 추가로 SMILE은 HDAC 중 하나인 sirtuin type 1을 끌어와, ERRγ와 직접 결합하여 표적유전자의 발현을 억제한다[12]. 하지만, SMILE과 HDAC의 상호작용에 의한 생리학적 효과는 아직 밝혀진 바가 없기 때문에 좀 더 많은 연구가 필요하다.
많은 핵수용체 및 bZIP 전사인자는 간에서 매개되는 내분비 대사 신호 조절에 관여한다. 포도당 대사는 주로 간에서 조절되는 대사 경로이다[30]. 포도당 대사에 관여하는 주요 보조활성화인자는 PGC-1α와 CREB-regulated transcription coactivator-2(CRTC-2)이다(Fig. 2). 이러한 보조활성인자는 섭식 및 공복 상태에서 췌장 β세포에서 분비되는 인슐린과 췌장 α세포에서 분비되는 글루카곤(glucagon)에 의해 조절된다[31-33]. 글루카곤은 공복 상태에서 G 단백질연결수용체(G protein-couped receptor)인 글루카곤 수용체에 결합하여 cAMP를 증가시켜 protein kinase A(PKA)를 활성화한다. 활성화된 PKA는 CREB을 인산화(phosphorylation)시켜 당신생합성 유전자의 발현을 증가시킨다[34]. 또한, 활성화된 CREB은 보조활성인자 CRTC-2와 상호작용하여 또다른 보조활성인자 PGC-1α의 발현을 증가시킨다[31,35,36]. 그 결과, PGC-1α은 핵수용체 HNF4α및 전사조절인자 FoxO1의 보조활성인자로 작용하여 당신생합성 유전자 발현을 유도한다[37,38]. 반면에, 섭식 상태에서 인슐린은 티로신 인산화효소 수용체(tyrosine kinase receptor)인 인슐린 수용체에 결합하여 인산화효소인 Akt를 활성화시킨다. Akt는 FoxO1을 인산화하며 동시에 salt-inducible kinase(SIK)을 활성화시키고, SIK은 CRTC-2를 인산화한다. 인산화된 CRTC-2와 FoxO1은 핵으로 이동하지 못해 글루카곤에 의해 조절되는 전사인자의 활성을 억제한다[39-41]. 이후에 마우스 모델 연구를 통해 인슐린에 의한 간의 당신생합성 억제가 보조억제인자인 SMILE의 활성과 관련이 있다는 것이 밝혀졌다(Fig. 2). 실제로, 인슐린은 인슐린/Akt 신호전달경로를 통하여 SMILE의 발현을 증가시켰다. 인슐린에 의해 유도된 SMILE 유전자 발현은 핵수용체 HNF4α와 보조활성인자 PGC-1α의 상호작용에서 PGC-1α와 경쟁하여 HNF4α에 의해 유도된 G6Pase 및 PEPCK와 같은 당신생합성 유전자의 발현을 감소시켰으며, 당뇨 모델 쥐에서 SMILE의 과발현은 고혈당을 개선시키는 결과를 보였다[14]. 추가로, SMILE은 CRTC-2와 경쟁을 통해 CREB/CRTC-2에 의한 PGC-1α와 PEPCK 발현을 억제하였다[42]. 이러한 결과는 SMILE이 보조억제인자로서 다른 보조활성인자와 경쟁하는 기전을 통해 포도당 대사를 조절하는 것을 알 수 있다. 한편, ERRγ 역시 포도당 대사를 조절하는 핵수용체로 알려져 있다. 공복 상태에서, ERRγ는 글루카곤/CREB/ CRTC-2 신호전달에 의해 발현이 증가하고, 보조활성인자인 PGC-1α와 복합체를 이루어 G6Pase 및 PEPCK 유전자의 발현을 조절한다고 알려졌다[43]. 또한, 보조억제인자인 SHP가 PGC-1α과 경쟁하는 기전을 통해 ERRγ에 의한 당신생합성을 억제하는 것을 확인하였다. 따라서, SMILE이 HNF4α와 PGC-1α의 상호작용에서 PGC-1α와 경쟁하여 HNF4α의 전사활성을 억제하기 때문에, ERRγ와 PGC-1α 간의 상호작용에 의한 전사활성도 SMILE이 조절할 수 있는지 추가 연구가 필요하다.
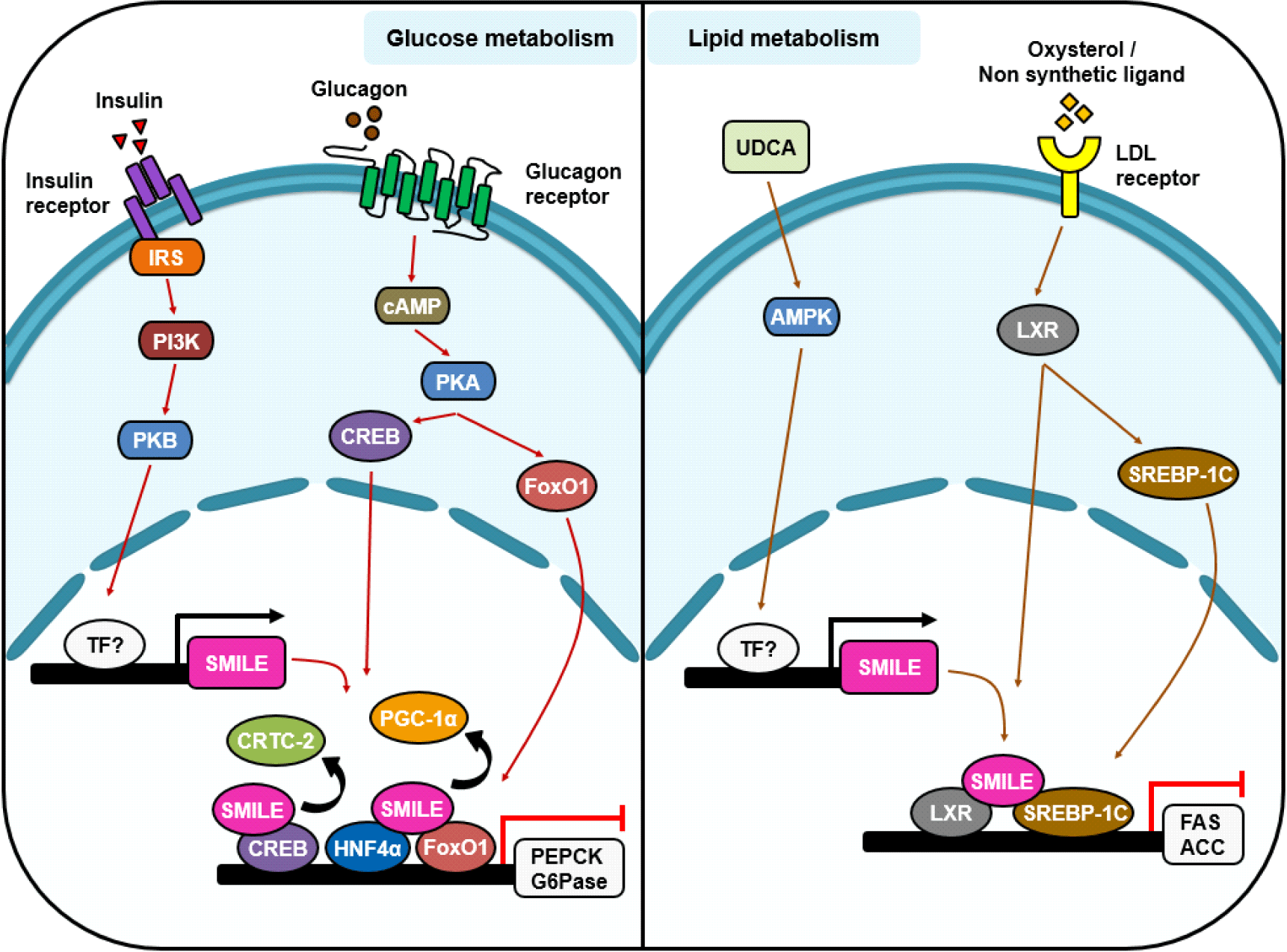
지질 대사에 관여하는 주요 전사 조절 인자는 LXR, SREBP-1c, PPARγ 및 ERRγ가 알려져 있으며 이들은 지방 생성 경로를 통해 조절된다[44-47]. LIPIN1은 지방 생성 단백질 phosphatidic acid phosphatase인 Lipin-1을 암호화하는 유전자이다. ERRγ와 Lipin-1에 대한 연구에서 ERRγ가 보조활성인자인 PGC-1α와 상호작용하여 Lipin-1의 발현을 유도하지만, SHP는 PGC-1α와 경쟁하여 Lipin-1의 발현을 억제한다[48]. SMILE 또한 SHP와 같은 보조억제인자이기 때문에, ERRγ의 전사 활성을 통해 Lipin-1과 관련된 지질 합성에도 관여할 수 있다. 지질 합성을 조절하는 SREBP-1c는 LXR과 콜레스테롤 부산물인 옥시스테롤을 통해 활성화된다[49,50]. SMILE은 LXR 및 SREBP-1c로 조절되는 지질 대사 속도제한효소(rate limiting enzyme)인 FAS(fatty acid synthase) 및 ACC(acetyl-CoA carboxylase)와 같은 표적유전자의 전사 활성을 억제하는 보조억제인자로 알려져 있다(Fig. 2). 또한, 우르솔산(ursolic acid, UA)은 LXR의 발현과 SREBP-1c의 프로모터 활성을 감소시키고 간 지질 함량을 감소시키는 식물 화합물이다. 흥미롭게도, 우르솔산은 간세포에서 AMP-activated protein kinase(AMPK) 인산화를 통해 SMILE 활성을 증가시킨다[51]. 이러한 결과는 우르솔산이 SMILE을 통해 SREBP-1C 및 LXR의 전사 활성 억제를 유도하는 잠재적 요인으로 작용할 수 있음을 나타낸다. 한편, SMILE은 인슐린/Akt 신호 경로를 통해 인슐린으로 매개되는 지방 생성을 조절한다. 인슐린에 의해 유도된 SMILE은 ATFs와 heterodimer를 이루어 insulin-induced gene-2(insig-2)의 발현을 감소시키고, 이러한 insig-2의 감소는 SREBP-1c에 의한 지방생성을 증가시킨다[52]. 추가로, SMILE의 간 특이적 제거(knockout)는 insig-2의 활성을 유도하고 지방 생성 과정을 억제하여 간 지방증을 약화시켰다. 흥미롭게도, SMILE은 인슐린 저항성 또는 ob/ob 쥐, 지방증이 있는 인간의 간에서 발현이 증가했다. 이러한 결과는 SMILE이 선택적 인슐린 저항성 하에 간에서 지속적인 지방 생성 발달에 잠재적 역할을 할 수 있으며, 나아가 지방간 질환 및 인슐린 저항성을 치료할 수 있는 표적유전자가 될 수 있다. 추가로, 간 지질 대사에서 SMILE의 기능에 대한 분석을 위해 중성 지방 항상성 및 대사에 중요한 담즙산에 초점을 맞추어 분석을 진행한 결과, 간 담즙을 유도하는 UDCA은 phosphoinositide 3-kinase/Akt/nuclear factor erythroid-2-related receptor 2 경로를 활성화하여 간 지방증과 인슐린 저항성이 개선되었음을 확인하였다. 한편, UDCA는 AMPK를 통해 SMILE의 발현을 증가시켰고, 이는 LXR과 상호작용하여 LXR로 매개된 지방 생성 유전자의 발현을 억제하였다[18]. 이러한 결과는 SMILE이 간 지방증, NAFLD 및 고지혈증과 같은 다양한 지질 대사 질환을 치료하기 위한 표적유전자가 될 수 있음을 시사한다.
결 론
SMILE은 bZIP 전사조절인자 중 하나인 CREB/ATF 계열 단백질이다. 그러나 기존 bZIP 단백질과 다르게 SMILE은 아스파라진 잔기의 결핍으로 DNA에 결합하지 못하지만 단백질과 상호작용이 가능하다. 따라서, SMILE은 전사조절인자인 핵수용체 혹은 bZIP 단백질과 상호작용하여 DNA 결합을 방해하거나, 보조활성인자와의 경쟁 혹은 직접적인 억제를 통해 다양한 대사과정에 관여하는 표적유전자의 발현을 조절한다. 특히, 이번 리뷰에서는 SMILE이 공동억제인자로서 간에서 중요한 당대사와 지질대사 조절을 통해 당뇨병, 고혈당 및 저혈당, 간지방증, 비알콜성지방간, 고지혈증과 같은 대사성질환을 제어할 수 있음을 보여주었다. 그러므로, SMILE은 다양한 대사와 관련된 질병의 치료제로서 중요한 표적유전자가 될 수 있음을 시사한다.






