서 론
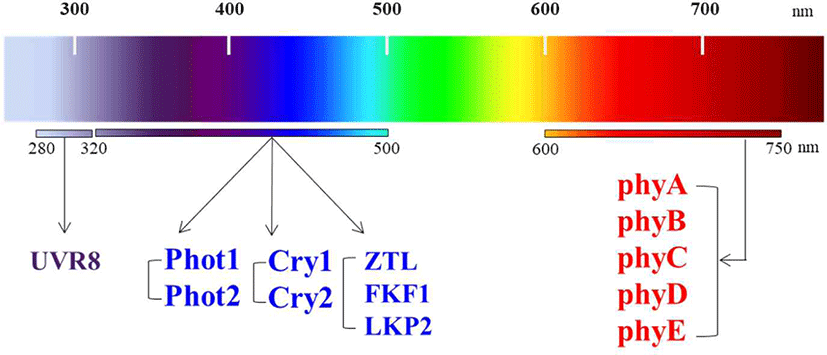
고착생물인 식물에게 빛은 필수적인 요소로서 광합성을 통한 생체에너지 확보와 더불어, 식물의 환경 적응 및 생존에 중요한 외부 환경변화 신호로 작용한다[1,2]. 눈이라는 시각 기관을 가져 이동성을 지닌 동물과는 달리, 이동성이 없는 식물은 시각 기관 대신 조직 전체에 환경신호로서의 빛을 인지할 수 있는 다양한 광수용체(photoreceptor)들을 지니고 있으며, 이들을 통해 고착된 조건의 척박한 환경에서도 분화 및 발달 조절이라는 방법을 통해 생존하고 있다[3,4]. 이렇게 식물이 빛 신호를 인지하고 반응하여 생장과 분화발달을 조절하는 것을 광형태형성(photomorphogenesis)이라고 한다. 현재까지 알려진 식물의 광수용체들로 파이토크롬(phytochrome), 크립토크롬(cryptochrome), 포토트로핀(phototropin), F-box를 함유하는 플라빈 결합 단백질(Zeitlupe [ZTL], Flavin-binding Kelch repeat F-box [FKF1], LOV Kelch repeat Protein 2 [LKP2]) 및 자외선 광수용체(UV resistance locus 8, UVR8) 등이 있다[5,6]. 식물 연구의 모델식물인 애기장대(Arabidopsis thaliana)에는 현재까지 총 13종의 광수용체들이 보고되었다(Fig. 1). 이들 광수용체들 중에서 파이토크롬(“plant color”에서 유래)에 대한 연구가 가장 활발히 이루어졌는데, 통상 쌍자엽 모델식물인 애기장대에는 5종의 파이토크롬(phyA−phyE)이 존재하는 반면, 단자엽 작물인 벼(Oryza sativa)에는 3종(phyA−phyC)이 존재하는 것으로 알려졌다[7,8]. 이 중에서 phyA는 빛에 의해 빠르게 분해되는 특징으로 Type I 파이토크롬으로 분류되며, phyB−phyE는 빛에 의한 분해가 매우 느려 Type II 파이토크롬으로 분류된다[9]. 또한 phyA는 원적색광 신호전달을 매개하고, phyB−phyE는 적색광 신호전달을 매개하는 것으로 알려졌다[10,11].
식물의 파이토크롬은 동일한 소단위(subunit) 2개가 결합된 이량체(dimer)로 존재하며, 소단위는 약 120 kDa 크기이며, 구형의 N-도메인(∼65 kDa)과 확장된 구조의 C-도메인(∼55 kDa)으로 구성된다[12,13]. 그리고 N-도메인 내 특정 cysteine 잔기에 phytochromobilin(PΦB)이라는 발색단이 thioether 공유결합으로 연결되어 있으며, 이를 통해 적색 및 원적색 빛을 흡수할 수 있는 색소 단백질이다. 통상 암(dark) 조건의 식물 내에서 파이토크롬은 적색광을 흡수할 수 있는 Pr 형태(red light-absorbing form)로 생합성 되며, 이후 적색광을 흡수하게 되면 원적색광을 흡수할 수 있는 Pfr 형태(far-red light-absorbing form)로 전환된다. 이때 파이토크롬이 Pfr 형태로 전환되는 조건에서 식물의 광 반응성과 생리학적 활성이 나타나기에, Pr은 불활성화 형태로, Pfr은 활성화 형태로 알려져 있다. 그리고 Pr 형태와 Pfr 형태는 광 조건에 따라 상호전환 가능하며, 이러한 광변환(photoconversion)은 파이토크롬 단백질 구조 변화를 유도하여 신호전달이 이루어지게 만드는 것으로 알려져 있다. 통상 파이토크롬 신호전달은 적색 또는 원적색이 포함된 광조건 하에서 만들어진 활성화 단백질 구조인 Pfr 형태가 핵으로 이동한 후 신호전달이 이루어지며, 궁극적으로 광형태형성 반응 유도를 위한 유전자 발현을 조절하게 된다.
파이토크롬은 종자 발아에서부터 잎과 줄기의 생장, 엽록체 발달, 광주기성(circadian rhythm), 음지회피성(shade avoidance), 개화(flowering) 등 식물의 거의 모든 생장과 발달 과정에 관여한다[11]. 파이토크롬 기능 분석을 위해 가장 많이 활용되고 있는 방법으로 유모(seedling)의 탈황화(deetiolation) 반응이 있다. 암 조건에서 식물이 발아하면 먼저 암형태형성(skotomorphogenesis) 반응을 보이는데, 이는 떡잎(cotyledon)이 닫힌 상태에서 정단 갈고리(apical hook) 형성과 함께 하배축(hypocotyl) 신장이 이루어지는 황화(etiolation) 표현형을 나타낸다. 이러한 암형태형성 반응은 흙 속에서 발아된 종자에서 만들어진 어린 식물이 흙을 뚫고 지표면으로 나오는 데 중요하다. 이후 식물이 빛에 노출되면 광형태형성 반응으로 정단 갈고리가 수직화 되면서 떡잎이 열리는 동시에 하배축 신장이 억제되는 탈황화 표현형이 나타나게 된다. 따라서 식물의 광 반응성 분석은 떡잎의 열린 각도나 하배축 길이 측정을 통해 이루어질 수 있다.
본 논문에서는 파이토크롬에 의한 식물의 광형태형성 조절기전을 통해 파이토크롬 광수용체에 의한 식물의 광신호전달 과정을 제시하고자 한다.
본 론
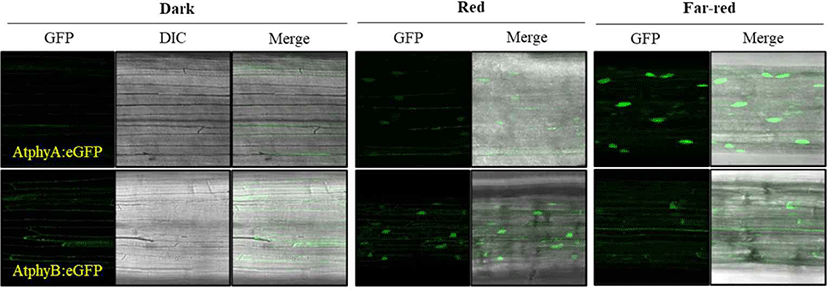
파이토크롬은 단백질 번역과정을 통해 세포질에서 생합성 되는데, 이때 엽록체(chloroplast)에서 생합성 되어 세포질로 나온 PΦB 발색단이 autocatalysis에 의해 결합되어 불활성화 Pr 형태로 만들어진다. 암 조건에서는 파이토크롬이 계속 생합성 되어 축적되면서 빛 신호에 민첩하게 반응하기 위한 대기상태를 유지한다. 실제로 암 조건에서의 식물 세포는 광형태형성 반응을 유도하기 위한 광수용체를 포함한 대부분의 필요 인자들을 보유하고 있는 상태이며, 빛이 들어와 광수용체가 인지하면 곧바로 반응이 시작될 수 있는 상태를 유지한다. 이에 파이토크롬에 의한 광신호전달의 첫 번째 단계는 세포질에 존재하는 Pr 형태의 파이토크롬이 빛을 인지하여 Pfr 형태로 전환되는 광활성화(photoactivation) 과정이다[14]. 적색 또는 원적색광이 함유된 빛이 들어오면 최소한 파이토크롬의 일부가 광활성화 되어 Pfr 형태로 전환되고, 이는 파이토크롬 단백질 형태 변화(conformational change)를 유도시켜 세포질에서 핵으로의 위치 이동이 진행되게 만든다[15,16]. 이때 phyA는 원적색광(far-red, FR) 하에서 핵으로 이동하며, phyB는 적색광 하에서 핵으로 이동한다(Fig. 2). 참고로 원적색광은 파이토크롬을 Pr 형태로 만들어 광활성화를 억제하는 조건이기에, 원적색광 하에서 phyA의 핵으로의 이동성은 한동안 설명이 되지 않는 난제였다. 이후의 연구를 통해, 적색광 조건에서는 phyA가 빠르게 분해된다는 특성과 원적색광 조건에서도 약 3% 정도의 phyA는 Pfr 형태를 지닌다는 점에서, 오직 원적색광 조건에서만 phyA는 핵으로 이동되어 축적될 수 있음이 증명되었다[10]. 즉, 적색광 하에서 phyA는 핵으로의 이동이 일어남과 동시에 단백질이 분해되는 기작으로 인해 phyA는 핵에 축적되지 못하며, 오직 단백질 분해가 억제되는 원적색광 하에서만 핵으로 이동된 phyA가 축적될 수 있기에, phyA는 원적색광 광신호전달을 매개할 수 있음이 제시되었다.
파이토크롬의 핵으로의 이동성에 대한 설명으로 phyB는 자체적으로 보유하고 있는 핵으로의 이동 신호(NLS, nuclear localization signal)를 이용하고, phyA는 자체 NLS를 보유하지 않아 핵으로의 이동을 돕는 FHY1(far-red elongated hypocotyl 1) 및 FHL(FHY1-like) 인자가 필요한 것으로 알려졌다[17]. 또한 FHY1/FHL 인자는 NLS뿐만 아니라, 핵으로부터 나오는 신호(NES, nuclear export signal)도 지니고 있으며, importin α(IMPα) 단백질이 FHY1/FHL 인자의 NLS 신호 서열을 인지하여 결합하면 핵으로 이동하는 것으로 확인되었다[18]. 따라서 phyA는 FHY1/FHL-IMPα 단백질 셔틀을 통해 핵으로 이동되는 것으로 여겨진다. 그에 반해, phyB의 경우 파이토크롬이 존재하지 않는 단세포 녹조류의 핵을 이용한 cell-free system 분석을 통해 자체 NLS가 아닌 PIF (phytochrome-interacting factor) 인자와 같은 수송 촉진제(transport facilitator)가 필요함이 제시되었다[19]. 따라서 phyB의 핵으로의 이동성에 대해서는 추가 연구가 필요할 것으로 여겨진다.
핵으로 이동하지 못하는 파이토크롬 돌연변이들은 식물의 광 반응성을 유도할 수 없음이 알려졌기에, 핵으로의 이동성은 파이토크롬 기능에 매우 중요하다[20]. 이때 핵으로의 이동성을 결정하는 핵심 요인은 파이토크롬의 광활성화 과정을 통한 Pfr 형태가 만들어지는 것이다. 기존 결과에 의하면 phyB의 자체 NLS가 Pr 형태에서는 감추어져 있다가 Pfr 형태가 만들어지면 노출됨으로써 핵으로 이동한다고 보고되기도 하였고[21], phyA는 Pfr 형태가 FHY1/FHL 인자와 특이적으로 상호작용하여 핵으로 이동하는 것이 보고되었다[22]. 그리고 수송 촉진제를 통한 핵으로의 이동성에서도 Pfr 형태만 PIF 인자와 상호작용하기에[19], 결론적으로 파이토크롬의 광활성화에 의한 Pfr 형태로의 구조 변화가 핵으로의 이동성에 필수적이라고 할 수 있다. 따라서 파이토크롬의 광에 의한 활성화와 핵으로의 이동은 신호전달의 최상위 단계를 구성하고 있으며, 파이토크롬이 기능하는 데 필수적인 역할을 담당하고 있다.
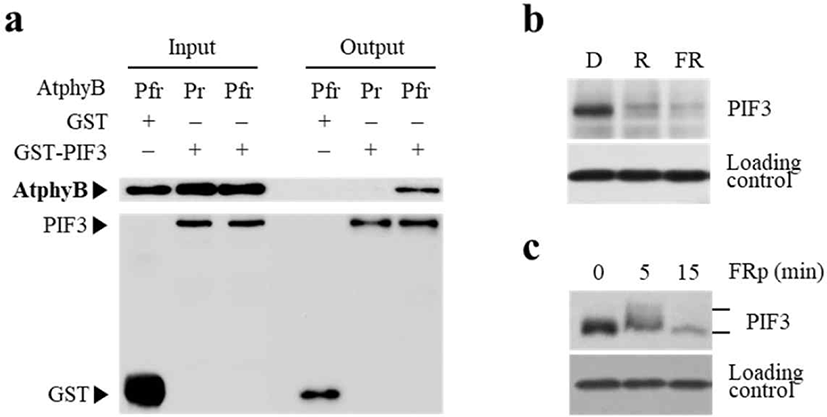
핵 내로 이동한 Pfr 형태의 파이토크롬은 다양한 하위 신호전달인자(downstream signal transducer)들과 상호 작용한다는 것이 잘 알려져 있다[23]. 현재까지 파이토크롬과 직접적으로 상호작용하여 광신호전달 네트워크를 구성하는 인자들만 수십 종에 달하며, 그 중에서 PIF 인자들이 파이토크롬 신호전달에서 가장 핵심적인 역할을 하는 것으로 잘 알려져 있다[24,25]. PIF 인자는 bHLH(basic helix-loop-helix) 전사인자로서 애기장대에 총 8종(PIF1−PIF8)이 존재하는 것으로 보고되었다[26]. 이들은 암 조건에서 광형태형성 반응을 억제시키면서 황화 표현형 등의 암형태형성 반응을 유지시키는 데 필요한 유전자들의 발현을 조절하는 것으로 보고되었다[27,28]. 파이토크롬은 PIF 인자와 Pfr 형태 특이적으로 상호작용하며(Fig. 3a), 암 조건의 식물체 내에 축적되어 작용하다가 빛이 조사되면 빠르게 분해되는 특징을 지니며(Fig. 3b), 이러한 광 의존적 단백질 분해 과정에서 인산화가 필요한 것으로 확인되었다(Fig. 3c). 현재까지의 연구로 파이토크롬은 PIF 인자의 DNA 결합을 방해하여 작용을 억제하고, 또한 PIF 인자가 인산화 된 후 식물 세포 내에 존재하는 ubiquitin 26S proteasome proteolytic pathway를 통해 분해되는 것을 촉진시키는 것으로 확인되었다[29,30]. 또한 파이토크롬은 단백질 키나아제(protein kinase) 활성을 지니고, PIF 인자를 직접적으로 인산화 시킬 수 있다는 증거도 제시되었다[31]. 따라서 파이토크롬은 광형태형성을 억제하는 PIF 인자를 인산화 시켜 분해시키는 방법으로 광신호전달을 매개하는 모델이 제시되었다[32].
또 다른 주요 파이토크롬 상호작용 인자들로 COP1(constitutively photomorphogenic 1) 및 SPA(suppressor of phyA-105) 단백질이 만드는 COP1-SPA complex가 있다. COP1은 식물뿐만 아니라, 동물에도 널리 존재하는 잘 보존된 E3 ubiquitin ligase 단백질이다[33]. SPA는 애기장대에 총 4종(SPA1−SPA4)이 존재하며, 파이토크롬 결핍 돌연변이를 정상으로 되돌리는 억제 돌연변이(suppressor) 연구를 통해 동정되었다. 이후의 연구를 통해 이들이 서로 상호작용하여 complex를 이루어 E3 ligase로 작동하여, 식물의 광형태형성 반응 조절의 master 전사인자로 알려진 HY5 (elongated hypocotyl 5) 등의 단백질을 유비퀴틴화시켜 26S proteasome 경로를 통해 분해시키는 것이 확인되었다[34,35]. 이때 Pfr 형태의 파이토크롬은 COP1-SPA complex에 결합하여 COP1-SPA 상호작용을 방해하여 complex를 해리시킬 수 있음이 증명되었다[36,37]. 즉, COP1-SPA complex는 광형태형성 반응의 주요 억제자로 역할을 하는데, Pfr 형태의 파이토크롬이 핵으로 들어와 이 complex와 상호 작용하게 되면 COP1-SPA 결합력이 약해지면서 complex가 해리되어, 궁극적으로 억제 기능을 발휘하지 못하게 됨으로써 식물의 광형태형성 반응이 유도되는 모델이 제시되었다[12,38]. 최근에는 파이토크롬-PIF-SPA complex 내의 단백질 키나아제 활성에 의한 인산화가 식물의 광형태형성 조절에 주요 역할을 한다는 것이 보고되기도 하였다[39].
고착생물인 식물은 환경 변화를 인지하여 분화발달을 조절하는 방식으로 생존하는데, 이때 유전자 발현 조절이 필연적으로 수반되기에, 이동성을 지닌 동물에 비해 식물의 반응성은 매우 느리게 진행된다. 식물이 광을 인지하고 반응을 나타내는데도 보통 짧게는 수 시간에서 며칠의 시간이 필요하다. 예로서, 암형태형성에서 광형태형성으로의 전환(i.e., deetiolation)을 조사하는 경우, 실험실에서는 하배축 길이를 측정하여 분석할 수 있는데, 일반적으로 광 처리 3–4일 이후에 측정한다. 이는 파이토크롬 광수용체가 광을 인지하여 핵으로 이동하고, 하위 신호전달인자들과의 상호작용을 통해 광 반응성을 나타내기 위한 유전자 발현을 조절해야, 궁극적으로 우리가 관찰할 수 있는 식물의 반응이 나타나기 때문이다. 이러한 과정은 광신호전달 네트워크뿐만 아니라, 식물 호르몬 신호전달 등 식물 세포 내 다양한 신호전달이 유기적으로 작동하면서 식물의 분화발달을 유도하기에, 상당한 시간이 소요된다. 애기장대 게놈 전체를 대상으로 한 발현 분석(genome-wide expression analysis) 결과에 따르면, 약 10%인 2,500개 유전자들이 파이토크롬 광활성화에 의해 발현이 변화하였으며, 이 중에서 약 80%는 발현이 증대된 반면, 약 20%는 발현이 감소된 것으로 나타났다[40–42]. 이들 유전자 대부분이 식물의 생장 및 분화발달과 밀접하게 연관된 것으로서, 광합성, 호르몬, 물질대사 등과 연관된 유전자들과 자가생장(autotrophic growth)에 필요한 유전자들이었다.
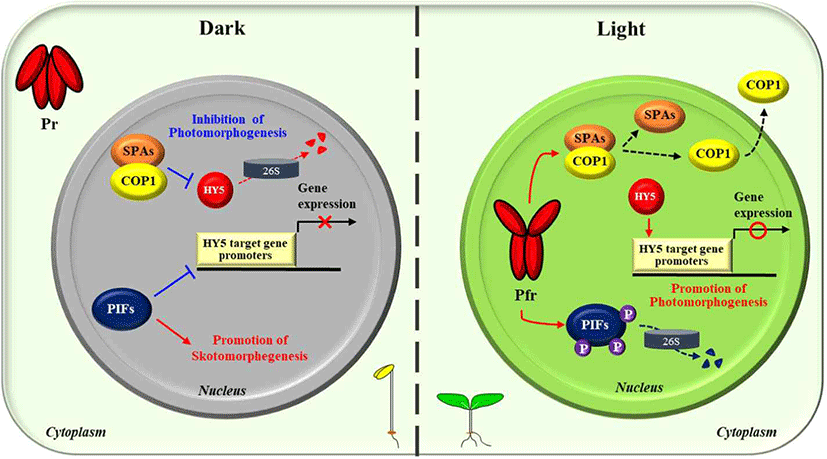
이러한 식물의 광형태형성 유도를 위한 광 반응성 유전자 발현 조절에서, 가장 핵심적인 역할을 하는 것이 bZIP 전사인자인 HY5이다[34,43]. 상기 유전자 발현의 대부분이 HY5 기능과 연관되어 있기에, HY5를 식물의 광신호전달 과정에 있어 master 전사인자로 칭하기도 한다. 즉, 암 조건의 식물 세포에서는 HY5 기능이 발휘되지 않도록 통제하여 광형태형성 반응을 억제해야 하고, 오직 광 조건에서만 HY5가 작동하도록 만들어야 한다. 이를 위하여 식물은 기본적으로 HY5 통제를 위한 최소 이중자물쇠를 채우고 있는데, 그 하나는 암 조건에서 축적된 PIF 인자가 HY5 전사인자가 작용하는 프로모터를 선점함으로써 HY5 기능을 제어하고 있고, 더 중요한 조절로 암 조건에서는 COP1-SPA complex가 HY5 유비퀴틴화시켜 26S proteasome에 의해 지속적인 분해가 이루어지게 만든다(Fig. 4). 이러한 전사(transcription) 및 번역 후 변형(post-translational modification) 수준에서의 이중 조절을 통해 암 조건에서는 HY5에 의한 유전자 발현 조절이 불가능하도록 만드는 것이다. 이러한 상황에서 빛이 조사되면, 제일 먼저 광수용체들이 광을 인지하게 되는데, 파이토크롬의 경우, Pfr 형태로 전환되어 우선적으로 핵으로 들어가, 한편으로는 PIF 인자를 인산화 시켜 분해되도록 만들고, 또다른 한편으로는 COP1-SPA complex를 해리시켜 불활성화를 유도하여, 궁극적으로는 광형태형성을 억제하고 있던 이중자물쇠를 해체시켜 광신호전달이 이루어지게 만든다(Fig. 4). 즉, 파이토크롬의 광활성화는 광형태형성 저해인자들인 PIF 인자 및 COP1-SPA complex를 불활성화시킴으로써, 광형태형성 촉진인자인 HY5 단백질 축적과 기능을 활성화시켜, 식물의 광 반응성 관련 유전자 발현이 전체적으로 이루어지게 만들어 광형태형성 분화발달이 진행되도록 만들게 된다.
결 론
식물의 광 반응성 또는 광형태형성 조절 과정에 대한 이해는 그동안의 활발한 연구와 함께 많은 연구결과들이 축적되면서 비약적으로 개선되었다. 파이토크롬 광수용체에 의한 식물의 광신호전달 체계도 상당히 확립되었는데, 현재까지의 연구결과를 토대로 간단하게 정리하면 크게 3 단계로 구분할 수 있다. 첫 번째 단계는 세포질에 존재하는 파이토크롬이 광에 의해 활성화 형태인 Pfr로 전환된 후 핵으로 이동하는 것이다. 두 번째 단계는 파이토크롬이 핵 내에서 다양한 하위 신호전달인자들과 상호작용하는 부분이며, 이 중에서 가장 중요한 것으로 광형태형성을 억제하는 PIF 인자의 인산화를 통한 단백질 분해 유도와 또다른 광형태형성 억제인자인 COP1-SPA complex를 해리시켜 불활성화 시키는 역할이다. 세 번째 단계는 파이토크롬-하위 신호전달인자의 상호작용의 결과로 광형태형성의 master 전사인자로 알려진 HY5 단백질 축적과 기능 활성화를 통해 HY5에 의한 광 반응성 유전자들의 발현 조절이 이루어지는 부분이다. 여기서 발현 조절되는 유전자들은 수천 개에 달하며, 광합성을 포함하여 식물이 자가영양체로 살아가는 데 필요한 유전자들과 더불어, 생장과 분화발달에 중요한 호르몬 및 물질대사 관련 유전자들이 포함된다. 이러한 유전자 발현 조절은 궁극적으로 다양한 환경에서 고착생물인 식물이 필요로 하는 적절한 분화발달을 유도하게 만들고, 이를 통해 다양한 환경조건에 따라 분화발달이 진행된 식물체들이 관찰될 수 있는 것이다. 이러한 식물의 광 반응성은 농업의 주요 형질의 하나인 농작물의 생산성과 밀접하게 연관되어 있다. 또한 최근 이산화탄소 등의 온실가스에 의한 지구온난화 문제가 크게 대두되고 있는데, 식물의 광 반응성 증대는 대기 중의 이산화탄소를 줄이는 데도 기여할 수도 있다. 따라서 본 연구주제는 향후 바이오에너지 생산에 필요한 바이오매스를 포함한 작물 생산성 향상과 이산화탄소 저감을 통한 지구온난화 문제를 개선하는 데 도움을 줄 수 있을 것으로 여겨진다.






