서 론
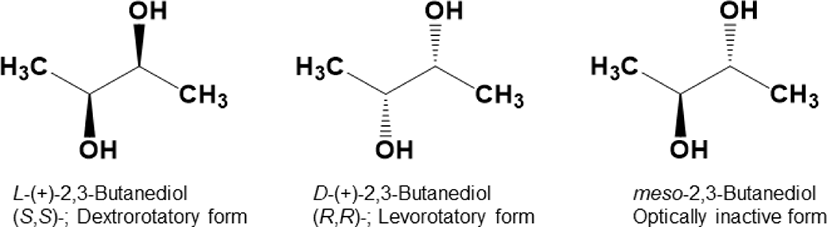
2,3-부탄다이올(2,3-Butanediol, 2,3-BDO)은 탄소수 4개로 구성되고 2번, 3번 탄소에 하이드록실기를 가지는 이중알코올(diol) 물질로(Fig. 1), 산업체에 주요 전구체로 널리 사용되고 있는 물질이다(Ji et al., 2011). 2,3-BDO의 수화를 통하여 합성고무의 원료인 1,3-부타디엔(1,3-butadiene)을 만들 수 있으며, 탈수소반응을 통하여 식품 산업에서 향기 성분으로 쓰이는 아세토인(acetoin)과 다이아세틸(diacetyl)을 만들 수 있다(Zeng and Sabra 2011). 2,3-BDO는 2,3-butene oxide의 가수분해를 통하여 화학적으로 합성할 수 있다. 하지만 이러한 방법은 가혹한 조건(160℃-220℃ and 50bar)에서 진행되어야 하며, butadiene, MEK, butenes과 같은 부산물을 생성하여 2,3-BDO를 정제하는데 추가 비용을 발생시킨다(Kim et al., 2017a).
따라서 산업에서는 주로 미생물 발효를 이용한 생물학적 방법을 통하여 2,3-BDO를 생산하고 있다(Bialkowska, 2016). 이때 Klebsiella pneumoniae, K. oxytoca, Enterobacter aerogenes, Paenibacillus polymyxa가 주로 이용된다. 이 미생물은 고수율, 고생산성으로 2,3-BDO를 생산할 수 있지만, 몇 가지 단점을 지닌다(Kim et al., 2017a). 첫째, Klebsiella spp.는 생물안전등급 2등급으로 분류되어 산업적 공정에 적용 시 많은 제약 및 규제를 받게 된다. 둘째, 대부분의 2,3-BDO 생산 박테리아는 광학이성질체인(2R,3R)-2,3-BDO, (2S,3S)-2,3-BDO, meso-2,3-BDO가 혼합된 형태를 생성하는데, 이는 산업적 활용의 제한사항이 될 수 있다.
최근 이에 대한 대안으로 효모인 Saccharomyces cerevisiae를 이용한 2,3-BDO 생산에 대한 연구가 활발히 진행되고 있다. S. cerevisiae는 GRAS(Generally Recognized As Safe) 균주로 예로부터 주정용, 제빵용으로 사용될 정도로 안전하며, 유전적 정보가 잘 알려져 있고, 유전자 도구가 많이 개발되어 있다. 또한 진핵세포로 박테리오파지에 대한 감염의 위험성이 없기 때문에 산업적 활용 가치가 높다. 이러한 장점에도 불구하고, 효모는 2,3-BDO를 거의 생산하지 못하기 때문에 2,3-BDO를 효율적으로 생산하기 위해서는 대사공학적으로 재설계된 S. cerevisiae의 구축이 필요하다(Kim et al., 2017a). 본 논문은 재조합 S. cerevisiae를 이용한 2,3-BDO의 생산에 관련된 최근 연구에 대하여 소개할 것이다. 첫째, 2,3-부탄다이올을 생산하기 위한 외래 경로의 도입, 둘째, 에탄올, 글리세롤과 같은 2,3-BDO 외의 부산물의 생성을 억제시키기 위한 대사공학적 접근 방법, 셋째, 경제성 확보를 위한 다양한 탄소원으로부터 2,3-BDO의 생산에 대하여 논의할 것이다.
본 론
S. cerevisiae에서 2,3-BDO 생합성에 관여하는 대부분의 효소는 세포질에 존재한다. 하지만 피루부산(pyruvate)을 α-acetolactate로 전환시키는 α-acetolactate synthase가 미토콘드리아에 존재하여 위치적으로 불리한 생합성 경로를 가지고 있다(Fig. 2). 또한 α-acetolactate를 acetoin으로 전환시키는 반응이 효소 반응이 아닌 자발적 반응에 의지하고 있어 생촉매 반응에 비해 비효율적이다. 이러한 문제점을 극복하기 위하여 외래 유래 효소인 α-acetolactate synthase, α-acetolactate decarboxylase를 도입하여 효율적으로 α-acetolactate를 acetoin으로 전환시킬 수 있는 생합성 경로가 구축되었다(Kim et al., 2017a). Bacillus subtilis 유래의 α-acetolactate synthase와 E. aerogenes의 α-acetolactate decarboxylase와 acetoin reductase를 도입하여 0.02 g/g의 수율로 2,3-BDO를 생산하였고(Ng et al., 2012), B. subtilis 유래의 α-acetolactate synthase와 α-acetolactate decarboxylase를 도입 및 S. cerevisiae 자체의 2,3-butanediol dehydrogenase의 과발현을 통하여 야생 S. cerevisiae의 생산량의 10배 이상의 2,3-BDO를 생산하였다(Kim et al., 2013).
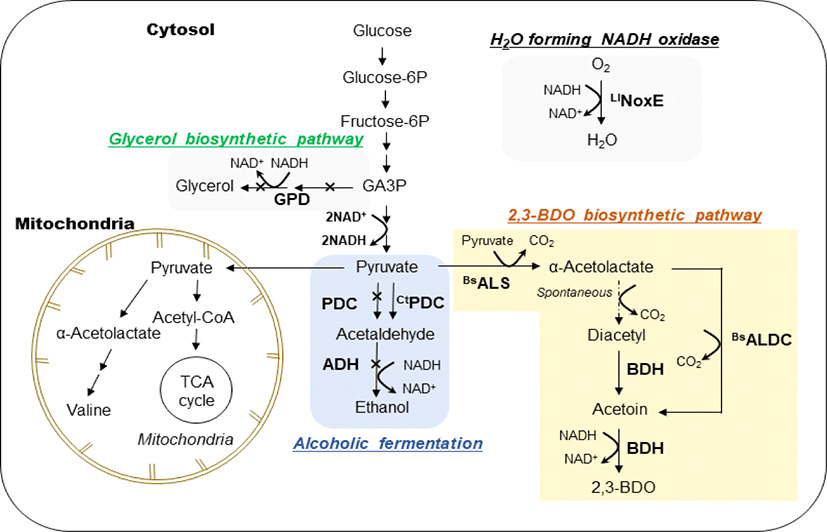
하지만 이러한 경우에도 S. cerevisiae는 주로 에탄올을 생성하고 상대적으로 적은 양의 2,3-BDO를 생산한다. 따라서 2,3-BDO 생산에 핵심 전구체인 피루부산이 에탄올로 전환되는 경로를 차단하여 피루부산을 축적시키고, 축적된 피루브산을 2,3-BDO로 효율적으로 전환시키는 생합성 경로의 재구축이 필요하다(Kim et al., 2017a). 피루브산으로부터 생성된 아세트알데하이드(acetaldehyde)를 에탄올로 전환시키는 효소 alcohol dehydrogenase를 암호화하는 ADH1, ADH3, ADH5를 파쇄하여 천연 S. cerevisiae 대비 47배 향상된 2,3-BDO 생산 수율(0.113 g/g)을 나타냈다(Ng et al., 2012). 또, 다른 연구에서는 ADH1의 파쇄와 함께 2,3-BDO 생합성 경로에 관여하는 효소의 집적화를 통하여 29.1 g/L의 2,3-BDO 생산량을 나타냈다(Kim and Hahn, 2014). 에탄올의 생성을 억제하기 위한 가장 효과적인 방법은 S. cerevisiae의 pyruvate decarboxylase(Pdc)의 활성을 제거하는 방법이다. 하지만 Pdc가 결여된 S. cerevisiae는 포도당을 대사하지 못하는 문제점을 가진다. 진화공법의 시행을 통하여 포도당 대사능이 개선된 S. cerevisiae를 선별하였고, 이 대사능 개선에 포도당 감지에 관여하는 전사 조절자를 암호화하는 MTH1 유전자의 돌연변이가 존재함을 밝혔다(Kim et al., 2013; Lian et al., 2014). Pdc가 불활성화된 S. cerevisiae에 2,3-BDO 생합성 경로 강화를 통하여 최종적으로 96.2 g/L의 2,3-BDO를 생산하였다(Kim et al., 2013).
S. cerevisiae는 포도당으로부터 피루브산 생성 시 두 분자의 NAD+를 NADH로 전환시킨다. 전환된 NADH는 피루브산이 에탄올로 전환되는 과정에서 NAD+로 재산화된다. 또, 다른 NADH 재산화 경로는 글리세롤 생산이며, 이러한 NADH 재산화를 통하여 S. cerevisiae는 효율적으로 포도당을 에탄올로 전환시킬 수 있다(Flikweert et al., 1996; Pronk et al., 1996). 하지만 대사공학적으로 재구축된 2,3-BDO 생합성 경로에서는 피루브산이 2,3-BDO로 전환될 때 한 분자의 NADH만 재산화되므로 세포 내에는 한 분자의 NADH가 잉여로 남게 되고, 이를 해결하기 위하여 에탄올 생합성 경로가 차단된 재조합 S. cerevisiae는 글리세롤을 과량 생산한다(Kim et al., 2013). 글리세롤의 과량 생산은 2,3-BDO 생산에 있어 생산 수율의 저하뿐만 아니라, 분리 정제 비용의 발생과 같은 문제점을 발생한다. 따라서 세포 내에 축적되는 NADH를 산화시킬 수 있는 다른 경로의 활성화가 필요하다.
가장 간단한 방법은 발효 조건 중 산소의 공급을 증가시켜, 세포의 호흡을 통하여 NADH를 재산화하는 방법이다. 산소 공급 농도를 증가시켜 글리세롤의 양을 줄이고, 2,3-BDO의 생산을 증대시킨 연구가 보고되었다. 하지만 과량의 산소 공급은 글리세롤의 축적을 감소시킬 수 있지만 2,3-BDO의 전구체인 acetoin이 쌓이는 문제점을 나타냈다(Kim et al., 2013). 따라서 이를 극복하고자 세포 성장기와 2,3-BDO 생산 시기를 산소 공급 농도로 구분하는 2단계 산소공급 시스템을 도입하였다. 예를 들면, 세포 성장 시에는 높은 산소 농도를 유지하며, 축적된 acetoin을 2,3-BDO로 전환할 때는 상대적으로 낮은 산소 농도를 유지하여 글리세롤의 축적을 74% 감소시켰다(Kim et al., 2016). 세포 내 NADH를 재산화시키는 두 번째 방법은 Lactococcus lactis 유래의 water-forming NADH oxidase(NoxE)를 S. cerevisiae 내에 도입하는 방법이다. 2,3-BDO 생합성 경로가 강화된 Pdc 결여 균주에 이 효소를 도입한 경우, 이 효소가 도입되지 않은 균주에 비해 2,3-BDO 수율이 24% 향상되었고, 글리세롤 축적이 65% 완화되었다(Kim et al., 2015). 하지만 글리세롤의 축적을 막는 가장 적극적인 방법은 글리세롤 생합성 경로를 차단하는 것이다. 글리세롤 생합성 경로의 차단과 NoxE 효소의 도입을 통하여 이론 수율 82%의 2,3-BDO를 1.43 g/L/h의 생산성으로 총 73 g/L 생산하였다(Kim and Hahn 2015). 마지막으로 흥미로운 NADH 재산화의 방법으로 Crabtree-negative 효모의 Pdc를 도입하는 것이다. S. cerevisiae와 같은 효모는 포도당에 의해 호흡에 관여한 효소의 발현이 억제되므로 산소 조건에서도 호흡 대신 에탄올 생성과 같은 발효과정을 통하여 해당과정에서 생성된 NADH를 재산화시킨다. 이러한 효모를 Crabtree-positive yeast라고 하며, Candida tropicalis, Kluyveromyces marxianus와 같은 crabtree-negative yeast에 비해 높은 Pdc 활성을 나타낸다(Flikweert et al., 1996; Pronk et al., 1996). 따라서 낮은 활성을 나타내는 C. tropicalis 유래의 Pdc를 Pdc가 결여된 S. cerevisiae에 도입하면 적정 농도의 에탄올을 생성하며, NADH를 재산화 시킬 수 있다. NoxE 도입 및 2단계 산소공급 시스템을 함께 도입하여 0.40 g/g의 2,3-BDO 수율과 1.98 g/L/h의 생산성으로 154.3 g/L의 2,3-BDO를 생산하였다(Kim et al., 2016).
최근 빠른 생장 속도를 보이며 혹독한 조건에서 내성을 지닌 산업용 polyploid S. cerevisiae를 모균주로 하여 2,3-BDO를 생산한 결과가 보고되었다(Lee and Seo, 2019). CRISPR-Cas9를 이용하여 polyploid S. cerevisiae의 에탄올 생성에 관여하는 PDC1, PDC6, ADH1 유전자를 파쇄하였고, 2,3-BDO 생합성 경로 및 NoxE를 과발현하여 최종적으로 178 g/L의 2,3-BDO를 생산하였고, 이는 현재 재조합 S. cerevisiae를 이용한 2,3-BDO 생산 공정 중 최대 생산량을 나타낸다.
저가의 폐자원(목질계, 해조류 바이오매스 등)으로부터 2,3-BDO를 생산하는 공정은 환경적, 경제적 의미에서 많은 장점을 가진다. 하지만 S. cerevisiae는 목질계 바이오매스에 다량으로 존재하는 자일로스를 대사하지 못하며, 해조류에 존재하는 갈락토스를 효율적으로 대사하지 못하는 단점을 가지고 있다. 따라서 이러한 탄소원을 효율적으로 이용하여 2,3-BDO를 생산하기 위해서는 S. cerevisiae 내 해당 탄소원의 대사 경로의 재구축이 수반되어야 한다.
재조합 S. cerevisiae를 이용하여 자일로스로부터 2,3-BDO를 생산하기 위하여, Scheffersomyces stipitis 유래의 xylose reductase(XR), xylitol dehydrogenase (XDH)와 S. cerevisiae의 xylulose kinase(XK)의 과발현을 통하여 자일로스로부터 피루부산을 축적하는 S. cerevisiae를 구축하였다. 이 균주에 추가적으로 2,3-BDO 대사경로를 도입하여 자일로스로부터 44 g/L의 2,3-BDO를 생산하였다(Kim et al., 2014). 추가적으로 돌연변이 XR, Tal1, L. lactis NoxE, C. tropicalis Pdc를 재조합 S. cerevisiae 내에 추가 과발현하여 0.58 g/L/h의 생산성으로 자일로스로부터 96.8 g/L의 2,3-BDO를 생산하였다(Kim et al., 2017b). 또한 cellobiose로부터 2,3-BDO를 생산한 연구가 보고되었다. Cellobiose transporter와 β-glucosidase가 2,3-BDO 생합성 경로와 함께 도입된 재조합 S. cerevisiae는 cellobiose를 기질로 사용하여 5.3 g/L의 2,3-BDO를 생산하였다(Nan et al., 2014).
Gelidium amansii와 같은 해조류 유래의 바이오매스에는 포도당 외에 갈락토스가 다량 존재하며, 이는 2,3-BDO 생산에 좋은 탄소원이 될 수 있다. 포도당과 갈락토스의 동시 대사를 위하여 돌연변이 Mth1을 과발현하였고, 2,3-BDO 생합성 경로를 도입하여 약 100 g/L의 2,3-BDO를 생산하였다(Lian et al., 2014). 포도당과 갈락토스 대사에 있어 두 탄소원의 비율은 중요한 요소이므로 최적의 탄소원 조건(60 g/L 포도당과 60 g/L 갈락토스)을 탐색하였고, 이 조건에서 돌연변이 Mth1을 가진 S. cerevisiae는 100 g/L의 2,3-BDO를 생산하였다(Choi et al., 2016).
산업적 활용을 위해서는 바이오매스 당화액을 이용한 2,3-BDO 생산 공정의 구축이 중요하다. BD5_T2nox_dGPD1,2_CtPDC1균주를 이용하여 121 g/L의 포도당을 함유한 Cassava 당화액을 탄소원으로 이용하는 유가식 배양을 통하여 47.4 g/L의 2,3-BDO를 생산하였다(Kim et al., 2019). 또한 산업용 균주를 이용하여 구축한 2,3-BDO 생산 S. cerevisiae는 Cassava 당화액로부터 최종 132 g/L의 2,3-BDO를 생산하였고, 이는 향후 재생가능한 바이오매스의 활용 측면에서 의의가 크다(Lee and Seo, 2019).
S. cerevisiae를 이용한 2,3-BDO 생산 공정은 박테리아를 이용한 공정과 달리 박테리오파지에 대한 감염의 위험성이 없으므로 공정상의 안정성을 제공한다. 또한 대부분의 박테리아가 2,3-BDO를 광학 이성질체의 혼합물로 생산하는 반면, 재조합 S. cerevisiae는 단일 형태의 광학이성질체를 선택적으로 생산할 수 있어 생산된 2,3-BDO는 향후 의약품과 같이 고부가가치의 물질로 활용될 수 있다(Kim et al., 2014). 하지만 세포 내 NADH 불균형으로 인한 글리세롤의 축적과 세포 성장 저해 현상을 조금 더 개선한다면, 재조합 S. cerevisiae를 이용한 2,3-BDO 생산공정은 산업적으로 충분히 활용될 수 있는 가치를 지닐 것으로 사료된다.
요 약
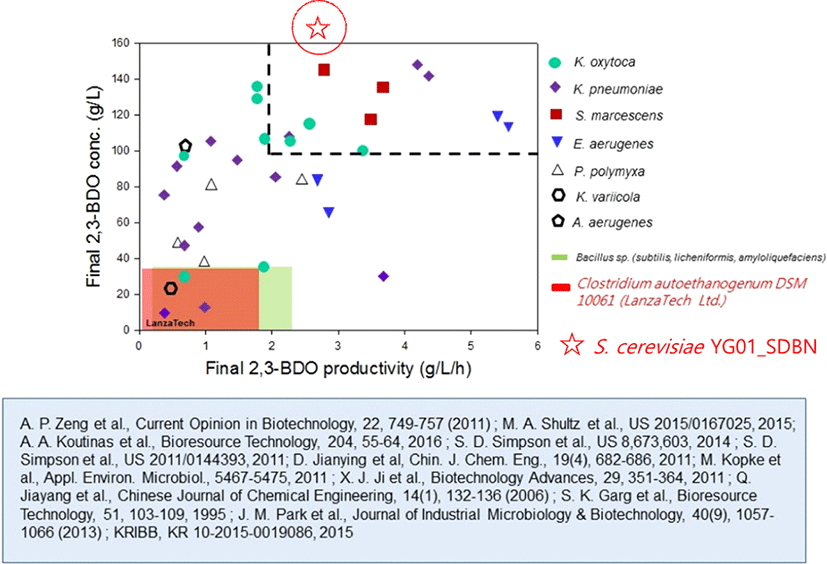
최근 몇 년간 재조합 S. cerevisiae를 이용한 2,3-BDO 생산 기술이 상당히 발전하였다(Table 1). 외래 유래의 2,3-BDO 생합성 경로 도입, 에탄올과 글리세롤 생합성 경로와 같은 경쟁 경로의 차단, 세포 내 NADH 불균형 해소를 위한 노력, 빠른 세포 성장을 보이는 산업용 S. cerevisiae의 사용 등을 통하여 재조합 S. cerevisiae는 2.64 g/L/h의 생산성으로 178 g/L의 2,3-BDO를 생산할 수 있다. 이러한 재조합 S. cerevisiae을 이용한 2,3-BDO 생산량은 2,3-BDO 생산 박테리아와 비교했을 때에도 충분한 경쟁력을 가진다(Fig. 3). GRAS 균주인 S. cerevisiae를 이용한 공정상의 장점이 충분히 존재하므로 재조합 S. cerevisiae를 이용한 2,3-BDO 생산공정은 향후 산업적으로 활용하기에 가치가 있을 것으로 판단된다.
| Mother strain | Genetic modification | Overexpressed genes | Culture | 2,3-BDO | Reference | |||
|---|---|---|---|---|---|---|---|---|
| Carbon source | Cluture mode | Productivity (g/L) | Yield (g/g) | Productivity (g/L/h) | ||||
| CEN.PK2-1C | ∆adh1 | BsalsS, BsalsD, ScBDH1 |
Glucose | Fed-batch | 29.1 | N.M. | 0.29 | [7] |
| CEN.PK2-1C | ∆adh1, ∆adh2, ∆adh3, ∆adh4, ∆adh5, ∆gpd1, ∆gpd2 |
BsalsS, BsalsD, ScBDH1, LlnoxE |
Glucose | Fed-batch | 72.9 | 0.41 | 1.43 | [13] |
| D452-2 | ∆pdc1, ∆pdc5, MTH1_A81P |
BsalsS, BsalsD, ScBDH1 |
Glucose | Fed-batch | 96.2 | 0.28 | 0.39 | [6] |
| D452-2 | ∆pdc1, ∆pdc5, MTH1_A81P |
BsalsS, EaalsD, ScBDH1 |
Glucose | Fed-batch | 132.4 | 0.34 | 0.41 | [18] |
| D452-2 | ∆pdc1, ∆pdc5, ∆pdc6 |
BsalsS, BsalsD, ScBDH1, LlnoxE |
Glucose | Batch | 29.4 | 0.37 | 0.40 | [12] |
| D452-2 | ∆pdc1, ∆pdc5, ∆pdc6 |
BsalsS, BsalsD, ScBDH1, LlnoxE, CtPDC1 |
Glucose | Fed-batch | 154.3 | 0.40 | 1.97 | [11] |
| D452-2 | ∆pdc1, ∆pdc5, ∆pdc6, ∆gpd1, ∆gpd2 |
BsalsS, BsalsD, ScBDH1 LlnoxE CtPDC1 |
Glucose | Fed-batch | 108.6 | 0.46 | 1.55 | [19] |
| JHS200 | ∆pdc1, ∆pdc6, ∆adh1 |
BsalsS, BsalsD, ScBDH1 LlnoxE |
Glucose | Fed-batch | 178.0 | 0.36 | 2.64 | [14] |
| D452-2 | ∆pdc1, ∆pdc5, MTH1_A81P |
BsalsS, BsalsD, ScBDH1, SsXYL1, SsXYL2, ScXYL3 |
Glucose, xylose | Fed-batch | 43.6 | 0.27 | 0.20 | [15] |
| D452-2 | ∆pdc1, ∆pdc5, |
BsalsS, BsalsD, ScBDH1, SsXYL1, SsXYL2, ScXYL3, SsXYL1mut, ScTAL1 LlnoxE CtPDC1 |
Glucose, xylose | fed-batch | 96.8 | N.M | 0.58 | [16] |
| D452-2 | ∆pdc1, ∆pdc5, MTH1_A81P |
BsalsS, BsalsD, Nccdt1, Ncgh1-1, |
Cellobiose | Batch | 5.3 | 0.29 | 0.22 | [17] |
| CEN.PK2-1C | ∆pdc1, ∆pdc5, ∆pdc6 |
SccytoILV2, BsalsD, ScBDH1, truncatedMTH1 |
Glucose, galactose | Fed-batch | 100 | N.M. | N.M. | [8] |
| D452-2 | ∆pdc1, ∆pdc5, ∆pdc6, ∆gpd1, ∆gpd2 |
BsalsS, BsalsD, ScBDH1, LlnoxE, CtPDC1 |
Cassava hydrolysate | Fed-batch | 47.4 | 0.39 | 1.03 | [19] |
| JHS200 | ∆pdc1, ∆pdc6, ∆adh1 |
BsalsS, BsalsD, ScBDH1, LlnoxE |
Cassava hydrolysate | Fed-batch | 132 | 0.32 | 1.92 | [14] |
* The superscripts in the enzyme names present as follows:
* Bs, B. subtilis; Ct, C. tropicalis; Ea, E. aerogenes; Ll, L. latis; Nc, N. crassa; Sc, S. cerevisiae; Sccyto, S. cerevisiae cytosolic; Ss, S. stipitis.
* Genes coding for enzymes as follows:
* adh1, adh2, adh3, adh4, and adh5 genes, alcohol dehydrogenase; gpd1 and gpd2 genes, glycerol-3-phosphate dehydrogenase; pdc1, pdc5, and pdc6 genes, pyruvate decarboxylase; alsS and ILV2 genes, α-acetolactate synthase; alsD gene, α-acetolactate decarboxylase; BDH1 gene, 2,3-BDO dehydrogenase; noxE gene, NADH oxidase; XYL1 gene, xylose reductase; XYL2 gene, xylitol dehydrogenase; XYL3 gene, xylulose kinase; TAL1 gene, transaldolase; cdt1 gene, cellodextrin transporter; gh1-1 gene, intracellular β-glucosidase; MTH1 gene, transcriptional regulator involved in the glucose-sensing signal transduction pathway.